- NUCLÉIQUES (ACIDES)
- NUCLÉIQUES (ACIDES)Découverts en 1868 par le biologiste suisse Friedrich Mischer dans les noyaux cellulaires, d’où leur nom (du latin nucleus , noyau), et également présents dans le cytoplasme, les acides nucléiques sont des molécules d’origine naturelle qui jouent un rôle fondamental dans la vie et la reproduction des cellules animales, végétales et microbiennes.Tels qu’on peut les isoler des tissus animaux ou végétaux, ces acides se présentent sous forme de molécules géantes ou de macromolécules, dont la masse moléculaire est comprise entre 25 000 et plusieurs centaines de millions. En effet, de même que les protéines sont formées par l’enchaînement de nombreux aminoacides et les polysaccharides par l’enchaînement de nombreuses molécules de sucres, les acides nucléiques sont constitués par l’enchaînement de nombreux motifs relativement simples, dissociables par hydrolyse; chacun d’eux comporte une base azotée (purique ou pyrimidique), un sucre à cinq atomes de carbone ou pentose (ribose ou désoxyribose) et un acide phosphorique: ce sont donc des esters phosphoriques complexes que l’on nomme nucléotides. La structure du pentose est à l’origine de la classification des acides nucléiques naturels en deux catégories fondamentales: d’une part, les acides ribonucléiques (ARN) contenant comme pentose le ribose ; d’autre part, les acides désoxyribonucléiques (ADN) contenant comme pentose le désoxyribose .L’histoire des acides nucléiques commença par des travaux sur la structure de leurs constituants moléculaires, puis elle évolua rapidement vers des travaux de génétique fondamentale qui mirent en évidence la fonction de continuité génétique qu’exercent les acides désoxyribonucléiques (et même les acides ribonucléiques chez certains virus). Ils renferment, en effet, dans la séquence de leurs nucléotides le patrimoine héréditaire de chaque individu et le code génétique permettant à chaque cellule de reproduire deux cellules filles en tous points identiques aux modèles parentaux. Les progrès remarquables des connaissances sur la structure et le rôle des acides nucléiques ont donné naissance à la biologie moléculaire, qui a pour but de rationaliser les données de la biologie descriptive classique en étudiant les processus de vie et de reproduction cellulaires au niveau des interactions entre molécules biologiques essentielles: acides nucléiques et protéines.1. NomenclatureNucléotidesLes bases azotées, dites nucléobases, qui entrent dans la constitution des nucléotides sont des bases organiques complexes dérivant de deux noyaux fondamentaux, la pyrimidine et la purine (tabl. 1). Le plus simple de ces deux noyaux, la pyrimidine, comporte deux atomes d’azote et quatre atomes de carbone, le tout formant un hétérocycle de six atomes. Le noyau de la purine est un hétérocycle comportant en tout neuf atomes: cinq de carbone et quatre d’azote. Les nucléobases puriques sont l’adénine et la guanine, les nucléobases pyrimidiques sont la cytosine, l’uracile (dans l’ARN) et la thymine (dans l’ADN).Les nucléosides sont des osides résultant de l’union d’une nucléobase avec un sucre à cinq atomes de carbone (pentose), qui est soit le ribose, ou 廓-D-ribofuranose, soit le désoxyribose ou 廓-D-2-désoxyribofuranose (fig. 1). Les nucléosides sont donc, selon le cas, des 廓-D-ribofuranosides ou des 廓-D-2-désoxyribofuranosides (fig. 1).Les nucléotides résultent de la phosphorylation des nucléosides. Dans le cas des mononucléotides qui existent à l’état libre dans les cellules vivantes, où leur rôle biochimique est fondamental, la position du groupement phosphoryle sur le sucre définit, pour une même base et pour un même sucre, deux nucléotides isomères différents dans le cas du désoxyribose, selon que le groupement phosphoryle est fixé en C-3 ou en C-5 et trois nucléotides isomères dans le cas du ribose selon que le groupement phosphoryle est fixé en C-2 , C-3 ou C-5 .Dans la figure 1 se trouvent indiquées les structures des principaux nucléotides que l’on peut obtenir par phosphorylation de l’adénosine ; les mêmes types de combinaisons sont possibles à partir de tous les autres nucléosides.PolynucléotidesLes polynucléotides sont des macromolécules constituées par l’enchaînement de plusieurs nucléotides reliés entre eux par une liaison 3 , 5 -phosphodiester: un seul groupement phosphoryle réunit les deux nucléotides contigus en estérifiant d’une part l’hydroxyle en position C-3 du nucléotide n et d’autre part le groupement hydroxyle en position C-5 du nucléotide n + 1. Ce type de liaison est le même en série désoxyribonucléique et en série ribonucléique (fig. 2).Le polynucléotide est par conséquent un polymère, constitué par une chaîne plus ou moins longue de monomères, les nucléotides. Or ces derniers sont de quatre sortes, si bien que leur enchaînement réalise une séquence dans laquelle l’ordre de succession des bases donne une identité particulière à chaque polynucléotide.Les acides désoxyribonucléiquesLes acides désoxyribonucléiques sont des polynucléotides constitués par l’enchaînement d’un grand nombre de mononucléotides du type:
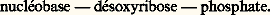 La base est soit de l’adénine, soit de la guanine, soit de la cytosine, soit de la thymine. On extrait facilement un ADN de haute masse moléculaire à partir du thymus de veau. La masse moléculaire de l’ADN isolé à partir de nombreuses sources, animales ou végétales, telles que thymus, germe de blé, bactéries, bactériophage, varie entre 1 million et plusieurs centaines de millions.Les acides ribonucléiquesLes acides ribonucléiques sont constitués par l’enchaînement d’un grand nombre de nucléotides du type:
La base est soit de l’adénine, soit de la guanine, soit de la cytosine, soit de la thymine. On extrait facilement un ADN de haute masse moléculaire à partir du thymus de veau. La masse moléculaire de l’ADN isolé à partir de nombreuses sources, animales ou végétales, telles que thymus, germe de blé, bactéries, bactériophage, varie entre 1 million et plusieurs centaines de millions.Les acides ribonucléiquesLes acides ribonucléiques sont constitués par l’enchaînement d’un grand nombre de nucléotides du type: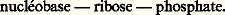 On trouve les mêmes bases que dans les ADN, à cette différence près que l’uracile remplace la thymine. Selon leur masse moléculaire et selon la fonction qu’ils assument, on distingue trois classes principales d’acides ribonucléiques:– Les acides ribonucléiques de transfert (ARN-t) sont des enchaînements de quatre-vingts nucléotides environ; ils doivent leur nom au fait qu’ils servent à transporter les acides aminés activés au cours de la synthèse des protéines. La structure primaire de plusieurs de ces molécules a été déterminée.– Les acides ribonucléiques «messagers» (ARN-m) résultent de la transcription de l’ADN par des enzymes spécifiques, les transcriptases, et constituent le code génétique utilisé par les ribosomes pour la synthèse des protéines. La masse moléculaire des ARN messagers est variable selon la longueur de la protéine qu’ils ont à synthétiser.– Divers acides ribonucléiques macromoléculaires jouent un rôle primordial: d’une part les ARN constitutifs des virus à ARN (virus de la mosaïque du tabac, de la grippe, de la poliomyélite, etc.); d’autre part, les ARN constitutifs des ribosomes, particules du cytoplasme cellulaire au niveau desquelles l’assemblage des aminoacides permet la biosynthèse des protéines.2. Structure des acides nucléiquesConformation et masse moléculaires des ARNLes ARN messagers sont des molécules filamenteuses à chaîne simple, donc monocaténaires, plus ou moins longues et de stabilité souvent précaire.Les ARN de transfert, dont on verra le rôle dans la dernière partie de cet article, sont constitués par l’enchaînement de quatre-vingts nucléotides environ, le tout ayant une masse moléculaire de l’ordre de 25 000. L’alanyl-ARN-t de levure représenté sur la figure 3 comporte soixante-dix-sept nucléotides et sa masse moléculaire est de 26 000.Dès que cette séquence et celles d’autres ARN de transfert furent connues, on se rendit compte qu’il existait une possibilité d’appariement entre les bases (stabilisant la structure de la molécule) par l’intermédiaire de liaisons non covalentes entre A 漣 U et G 漣 C (mais à l’encontre de l’ADN, le couple A 漣 U au lieu du couple A 漣 T). Plusieurs modèles ont été proposés, mais il semble que le modèle «en feuille de trèfle» rende le mieux compte des résultats expérimentaux. Des modèles de structure à trois dimensions (structure tertiaire) ont été prévus.D’autres ARN, à structure plus complexe que les ARN de transfert, c’est-à-dire par exemple l’ARN 5 S d’Escherichia coli , les ARN constitutifs des ribosomes, les ARN viraux, font également l’objet d’études assidues.Conformation et masse moléculaires de l’ADNL’ADN isolé à partir d’une source naturelle comme le thymus de veau se présente sous forme de fibres blanches et légères, comparables à première vue à de la cellulose. Sous forme de sel de sodium, l’ADN se dissout lentement dans l’eau pour former des solutions colloïdales extrêmement visqueuses.Depuis le début de ce siècle, on sait que l’ADN contient quatre bases principales: adénine (A), cytosine (C), guanine (G) et thymine (T). Le dosage de ces bases par les anciennes méthodes fit croire à leur répartition équimoléculaire, d’où la célèbre théorie des tétranucléotides qui prévalut pendant longtemps et ne fut réfutée que vers les années 1948-1950 lorsque l’on sut doser avec plus de précision les purines et les pyrimidines. Ce fut l’application extensive de la chromatographie sur papier, notamment par E. Chargaff et ses élèves, qui permit de définir un véritable atlas de la constitution en nucléotides d’un grand nombre d’acides désoxyribonucléiques naturels.La connaissance de la constitution en bases d’un ADN permet de déterminer la constitution globale de cet acide nucléique, puisque l’on sait par définition que chaque nucléotide comporte une base (à choisir parmi quatre), un pentose qui est toujours le désoxyribose et un groupement phosphoryle .Une constatation fondamentale devait se faire jour progressivement: d’une part, l’égalité des proportions en adénine et thymine et en guanine et cytosine; la constance du rapport (A + T)/(G + C) pour une même espèce animale, d’autre part. Cette constatation a pour corollaire immédiat que le rapport purines/pyrimidines est toujours égal à 1.La nature de la liaison entre nucléotides fut longtemps controversée, mais, sur la foi d’études aux rayons X et d’études enzymatiques, on s’est arrêté finalement à la liaison 3 -5 : comme on l’a déjà vu, le groupement phosphoryle établit un pont entre le groupement hydroxyle en position C-3 d’un premier nucléotide et le groupement hydroxyle en position C-5 d’un second nucléotide (fig. 2).Compte tenu des relations AT-GC, de la liaison 3 -5 -phosphodiester et des clichés obtenus aux rayons X, J. D. Watson et F. H. C. Crick proposèrent en 1952-1953 un schéma général de la conformation spatiale de l’ADN; ce schéma, mettant en évidence le rôle génétique de cet acide nucléique, est à la base de la théorie de Watson et Crick; il constitue l’un des plus grands événements scientifiques de ce siècle (fig. 4 a; cf. MA- CROMOLÉCULES).On doit se représenter la molécule d’ADN sous forme de deux chaînes hélicoïdales enroulées autour d’un même axe et maintenues sous forme d’une structure à deux brins ou structure bicaténaire (du latin catena , chaîne) grâce à l’«appariement» des bases se faisant vis-à-vis sur l’une et l’autre chaîne (fig. 4): les appariements sont toujours adénine-thymine (deux liaisons non covalentes ou liaisons hydrogène) et guanine-cytosine (trois liaisons non covalentes).Cette structure qui réunit deux chaînes réciproquement complémentaires à l’égard de leurs séquences nucléotidiques est compatible avec la réplication de la molécule d’ADN (cf. infra ).On voit sur la figure 4 b que les deux chaînes ont une polarité opposée: ce sont des chaînes antiparallèles ; de telle sorte que les extrémités 3 et 5 sont inversement placées dans chacun des deux brins. La complémentarité séquentielle et l’antiparallélisme des deux chaînes expriment le fait que leur structure est réciproquement codée .Les mesures effectuées sur la masse moléculaire des ADN ont donné lieu à de nombreuses controverses et, malgré l’énormité du travail effectué, on ne peut considérer comme valables que les mesures relatives à des échantillons dont on est sûr que l’intégrité moléculaire a été respectée. Or, une telle certitude n’est obtenue que dans les cas où l’on peut constater de visu au microscope électronique que les molécules étudiées ne sont pas rompues.Au cours des temps, diverses méthodes ont été expérimentées avec plus ou moins de succès: diffusion, diffraction de la lumière, viscosité, sédimentation. En principe, la méthode qui offre la plus grande sécurité est celle qui consiste à mesurer effectivement la longueur de molécules entières sur un cliché agrandi pris au microscope électronique (fig. 5). Connaissant d’une part l’agrandissement et sachant d’autre part que l’unité de masse moléculaire par unité de longueur pour la chaîne de l’ADN est 1,92 憐 106 par nanomètre, la masse moléculaire totale peut aisément être établie par un calcul simple, sachant que l’écart entre deux nucléotides consécutifs est de 0,34 nm. L’ultracentrifugation, effectuée sur des molécules natives, permet aussi d’obtenir d’assez bons résultats, et confirme ainsi les données précédentes.On sait actuellement que la masse moléculaire de l’ADN monocaténaire du bactériophage 淋X-174 est de 1,6 憐 106, ce qui est une très petite masse moléculaire pour un ADN contenant toute l’information nécessaire à la réplication d’un micro-organisme entier. En effet, l’ADN du virus de la vaccine comporte 240 000 paires de nucléotides, il a une masse moléculaire de 157 憐 106. Quant à l’ADN d’un micro-organisme très connu comme E. coli , il comporte 3 400 000 paires de bases représentant une masse moléculaire de 2,3 憐 109.3. Synthèse totale des acides nucléiquesLes principes fondamentaux des synthèses de nucléosides et nucléotides furent décrits par Todd et ses collaborateurs avec pour point culminant la synthèse totale de l’adénosine triphosphate, ATP en 1948. La synthèse de dinucléotides, donc l’obtention contrôlée de la liaison internucléotidique 3 , 5 -phosphodiester devait suivre grâce aux efforts indépendants de l’école de Todd et de celle de Khorana au début des années cinquante. Le perfectionnement des techniques de synthèse des oligonucléotides, couplé avec l’utilisation des méthodes enzymatiques, principalement les ADN-ligases, devait aboutir en 1976 à la synthèse totale du gène de structure régissant la biosynthèse du tyrosyl-t-ARN et en 1981 à la synthèse totale du gène interféron.Si l’application des méthodes de synthèse de l’ADN nécessite une instrumentation et une habileté exceptionnelles, en revanche les principes fondamentaux qui les régissent sont relativement simples: dans un premier temps on effectue la synthèse chimique totale d’oligonucléotides pouvant comporter de dix à quinze chaînons, et, dans un second temps, on lie entre eux ces fragments, dans l’ordre obligatoire de la séquence recherchée, grâce aux ADN-ligases; une enzyme bien connue comme l’ADN-polymérase-1 de E. coli est capable d’effectuer cette liaison.Synthèse des oligonucléotidesPlusieurs méthodes ont été successivement décrites pour l’assemblage des nucléotides par la formation réussie de la liaison 3 , 5 -phosphodiester: la méthode phosphate diester; la méthode phosphate triester; la méthode phosphite triester.Selon la méthode phosphite triester qui semble la plus prometteuse à l’heure actuelle, on effectue la liaison 3 , 5 -phosphodiester en faisant réagir deux nucléosides convenablement protégés avec un dichloroalkyl-phosphite (fig. 6 a). Le dinucléoside monophosphite obtenu est oxydé par l’iode en solution aqueuse pour donner le dinucléoside monophosphate correspondant. Cette méthode représente un progrès appréciable par rapport aux méthodes antérieures utilisant des phosphates substitués, en particulier quant à la rapidité.Un autre progrès récent dans la synthèse des oligonucléotides réside dans la transposition de la méthode sur support solide. Le principe de cette méthode est le suivant (fig. 6 b): la première unité A de la chaîne est attachée à un matériel macroscopique tel que du polystyrène ou des billes de silice. La seconde unité B et tous les agents de condensation sont dissous dans un solvant approprié et mélangés avec le support solide. La réaction de condensation se produit entre B et A sur le support solide et la chaîne se trouve étendue à S 漣 A 漣 B. À la fin de la période de réaction, le surplus des réactifs et les solvants sont éliminés par lavage. On renouvelle l’opération autant de fois que le nécessite l’élongation de la chaîne. Avec une telle méthode, il est impératif que les rendements soient aussi près que possible de 100 p. 100 à chacune des étapes, sinon le rendement final après dix ou quinze étapes chute de façon verticale: à titre d’exemple, si le rendement de condensation à chaque étape est de 90 p. 100, au bout de dix étapes le rendement final n’est que de 35 p. 100.Cette méthode est automatisée, de telle sorte que la synthèse d’oligonucléotides comportant jusqu’à quatorze unités nucléotidiques soit maintenant un procédé de routine. La cadence est de l’ordre de 30 minutes par chaînon; le système ne nécessite pas d’expérience préalable de la part de l’utilisateur; il fonctionne jour et nuit sans avoir besoin de surveillance, jusqu’à la terminaison du programme qui a été introduit au début de l’expérience.De plus, le même principe s’applique aussi à la synthèse de l’ARN!...Assemblage des oligonucléotides et obtention de gènes synthétiquesL’assemblage des oligonucléotides obtenus par synthèse chimique totale est réalisé par liaison enzymatique au moyen d’ADN-ligases. Dans un exemple tel que celui représenté par la figure 6, on voit que deux icosanucléotides partiellement appariés forment un assemblage bicaténaire suffisamment rigide pour que l’on puisse y accrocher, grâce à une ADN-ligase, un oligonucléotide de la même longueur que les deux premiers et qui viendra lui-même s’apparier sur une partie de sa longueur. L’allongement progressif de la chaîne se fait donc par accrochage successif d’oligonucléotides synthétiques de séquence connue.L’une des performances les plus remarquables dans ce domaine est la synthèse totale du gène de l’interféron leucocytaire humain IFN- 見-1 par Edge et ses collaborateurs (1981). L’interféron IFN- 見-1 est une protéine comportant 166 résidus d’aminoacides. Ce gène exprimé par E. coli programme la synthèse d’un interféron en tous points identique au produit naturel (De Maeyer et al., 1982). Il s’agit d’un fragment d’ADN bicaténaire comportant 514 paires de nucléotides. La synthèse a comporté la fabrication préliminaire de 66 oligonucléotides dont la dimension s’échelonne entre 14 et 21 résidus, plus un dodécanucléotide, le tout par des procédés en phase solide. La liaison de ces oligonucléotides fut obtenue au moyen de l’ADN-ligase du phage T 4.Mais bien entendu, pour pouvoir reconstituer des molécules biologiquement actives, il est indispensable de connaître préalablement la séquence selon laquelle l’assemblage des nucléotides doit être réalisé: c’est cette analyse que nous exposerons maintenant.Détermination des séquences et de la structure primaire de l’ADNLa détermination de la séquence complète des nucléotides constitutifs d’ADN macromoléculaires biologiquement actifs représente une des performances les plus remarquables de ces dernières années. Un premier succès important fut remporté par Sanger et ses collaborateurs (1975) avec la détermination de la séquence complète des nucléotides constitutifs de l’ADN monocaténaire circulaire du phage 淋X-174, soit 5 375 nucléotides. Par la suite ces mêmes auteurs devaient perfectionner leur méthode par l’emploi d’inhibiteurs dits «terminateurs de chaînes» ce qui leur a permis de déterminer la séquence complète de l’ADN bicaténaire d’un organisme supérieur: il s’agit en fait du génome mitochondrial humain, soit 16 569 paires de bases (Anderson et al., 1981). Ces travaux considérables devaient valoir à Sanger l’attribution du prix Nobel de chimie, pour la seconde fois, en 1980.Méthode de SangerLe principe de cette analyse est le suivant: dans un premier temps on coupe l’ADN à étudier en certains points grâce aux enzymes adéquates, les nucléases, qui se caractérisent par leur remarquable spécificité d’action (tabl. 2), notamment les endonucléases de restriction [cf. GÉNIE GÉNÉTIQUE].On obtient de cette façon un certain nombre de fragments dits oligonucléotidiques; on sépare, et on purifie, ces fragments par électrophorèse sur gel de polyacrylamide. On détermine la séquence de chacun des fragments et on reconstitue la séquence originelle en juxtaposant tous les segments dûment identifiés.La découverte de nucléotides substitutifs dits terminateurs de chaîne a permis de séquencer les fragments d’ADN avec une grande sûreté. À partir d’un fragment monocaténaire dit chaîne 漣, qui jouera le rôle de matrice , on induit la synthèse d’une chaîne complémentaire +, sous l’action de l’ADN-polymérase-1, mais on peut bloquer l’allongement (élongation) de cette chaîne + grâce aux terminateurs.Le principe du fonctionnement des terminateurs de chaîne est illustré (fig. 7) par l’exemple du 2 , 3 -didéoxythymidine-5 -triphosphate (ddTTP) sur l’ADN-polymérase-1 lorsque ce composé est incorporé dans la chaîne nucléotidique croissante au lieu et place de l’acide thymidylique. L’absence sur ce ddTT du groupement hydroxyle en position C-3 stoppe l’élongation de la chaîne polynucléotidique à partir de l’endroit où normalement T aurait dû être incorporé. Si l’on incube une amorce (un court fragment d’ADN nécessaire pour amorcer la réaction de synthèse) avec l’ADN-polymérase-1 et une matrice (ADN monocaténaire inconnu) avec un mélange de ddTTP, de TTP (thymidine triphosphate normal) et des trois autres nucléosides triphosphates normaux dont l’un est marqué avec 32 P, l’ADN-polymérase catalyse la formation d’une série de chaînes complémentaires de la matrice; on obtient finalement un mélange de polynucléotides ayant tous la même séquence initiale et terminés par des groupements ddT à l’extrémité C-3 . Lorsque l’on fractionne ce mélange par électrophorèse sur gel de polyacrylamide, le diagramme de résolution montre toutes les bandes indiquant la distribution de ddT (fig. 8).En utilisant des terminateurs du même type pour les autres nucléotides dans des mélanges d’incubation séparés et en mettant les incubats en parallèle sur le gel d’électrophorèse, on obtient un diagramme de résolution dont on peut lire la séquence «comme dans un livre».On peut utiliser deux types de triphosphates terminateurs, les dérivés didésoxy et les arabinosides. Les ara-nucléoside-triphosphates fonctionnent comme des terminateurs de chaînes inhibiteurs de l’ADN-polymérase-1 de E. coli d’une façon comparable à ddTP bien que les chaînes se terminant par ara-C puissent être étendues par certaines ADN-polymérases de mammifères (cette propriété explique la valeur thérapeutique des arabinosides qui bloquent, par exemple, la réplication des ADN viraux en respectant la réplication des ADN des cellules hôtes: cf. THÉRAPEUTIQUE - Chimiothérapie). Afin d’avoir une résolution suffisante des bandes permettant une lecture de séquences étendues, il est obligatoire que la proportion de triphosphate terminateur par rapport au triphosphate normal soit telle que l’on ait une incorporation partielle du terminateur: il est évident que si l’on avait 100 p. 100 de terminateur, toutes les chaînes se termineraient avec l’incorporation de la première molécule d’analogue.Pour les didésoxy-nucléoside-triphosphates, la proportion est de 1 pour 100. Pour les ara-nucléoside-triphosphates, la proportion est de 1 pour 5 000.4. Propriétés physico-chimiquesUtilisation des radiations dans l’analyse des acides nucléiquesLes acides nucléiques présentent en solution diluée un très intense spectre d’absorption en ultraviolet ; ce spectre est la résultante des spectres d’absorption individuels des purines et pyrimidines entrant dans leur constitution. Du point de vue quantitatif, il est intéressant car il permet de doser les acides nucléiques en solution pure (fig. 9).Les méthodes utilisant la diffraction des rayons X fournissent des informations capitales en ce qui concerne la structure des molécules possédant un degré d’organisation élevé, ce qui est le cas des macromolécules d’intérêt biologique telles que les protéines cristallisées, les acides nucléiques macromoléculaires, les virus. Toutefois, la visualisation des structures à trois dimensions n’est pas directe et ne peut être obtenue que par une interprétation laborieuse des clichés photographiques de diffraction. On trouvera dans l’article BIOLOGIE MOLÉCULAIRE l’histoire de la découverte de la structure tridimensionnelle de l’ADN par W. T. Atsbury puis Wilkins, Watson et Crick.En revanche, la microscopie électronique permet une observation directe des macromolécules convenablement préparées; bien qu’elle nécessite un appareil expérimental onéreux et des spécialistes hautement qualifiés, cette technique est cependant d’une interprétation beaucoup plus accessible.Dans le cas des acides nucléiques, on doit d’abord isoler les molécules dans un état aussi voisin que possible de l’état natif pour éviter les fragmentations, puis les fixer par un ombrage métallique; on obtient alors des clichés très intéressants, qui ont permis non seulement d’avoir une idée de la conformation des acides nucléiques macromoléculaires, mais encore de mesurer la dimension exacte des molécules (fig. 10).CentrifugationL’ADN peut être fractionné par centrifugation en gradient de chlorure de césium, CsCl. Le césium a un poids atomique très élevé: 133. Si l’on soumet une solution de chlorure de césium 8 M à centrifugation rapide pendant trois jours, on constate qu’il s’établit un gradient linéaire de concentration correspondant à un gradient de densité entre 1,55 g/cm3 au sommet du tube et 1,80 g/cm3 au fond du tube. On peut dissoudre un mélange d’ADN dans la solution de chlorure de césium et centrifuger. Après soixante-douze heures, les différents ADN formeront des bandes à différents niveaux. On dit que la densité apparente d’une molécule d’ADN est la densité de chlorure de césium au niveau duquel l’ADN sédimentera en fin de centrifugation à l’équilibre. La densité apparente d’un ADN est proportionnelle au pourcentage de G-C qui le compose. On peut ainsi séparer différents ADN. On a également utilisé cette méthode pour séparer un ADN préalablement marqué par des isotopes lourds (15N, 2H) du même ADN non marqué. Les ARN extraits d’une cellule peuvent être fractionnés par centrifugation dans un gradient de saccharose linéaire entre 5 et 20 p. 100. Ce gradient est préformé dans le tube à centrifuger et on dépose à la surface le mélange d’ARN en solution. Après dix-huit heures de centrifugation à 50 000 g , on retrouve la plupart des ARN séparés (on les détecte en expulsant lentement le liquide du tube et en mesurant en continu la densité optique à 260 nm). Cette méthode peu résolutive permet de séparer les grands ARN ribosomaux et les ARN de transfert 4 S (fig. 10).ÉlectrophorèseL’électrophorèse en gel de polyacrylamide permet de séparer les macromolécules en fonction de leur charge et de leur poids moléculaire. Les acides nucléiques sont des polyanions comprenant la même charge par nucléotide, la séparation se fera seulement en fonction de leur longueur (et dans une certaine mesure de leur conformation).Nous avons vu l’application de cette méthode à la détermination de la séquence de l’ADN, sachant que la vitesse de migration électrophorétique de chaque fragment est inversement proportionnelle au logarithme de sa longueur.La même méthode appliquée à un mélange d’ARN permet de séparer les ARN 28 S, 18 S, 5 S, 4 S ainsi que dans certains cas les ARN messagers, lorsque ceux-ci sont suffisamment abondants et de taille homogène (fig. 10).5. Dénaturation de l’ADN. Hybrides moléculairesLorsque l’on chauffe une solution d’ADN, on constate que son absorption d’ultraviolet à 260 nanomètres augmente de 40 p. 100 à partir d’une certaine température, c’est l’effet hyperchromique . La température correspondant à la moitié de l’effet hyperchromique est appelée température de fusion ou Tm. L’effet hyperchromique correspond à une rupture des liaisons hydrogène entre les deux chaînes d’ADN. La séparation entre les deux chaînes avec perte de la structure secondaire de l’ADN est appelée dénaturation thermique de l’ADN (fig. 11).On peut par un traitement mécanique casser l’ADN en des fragments de longueurs plus ou moins homogènes. On chauffe le mélange au-dessus de la température de fusion, puis on refroidit progressivement. On constate alors que les chaînes complémentaires se réassocient en reformant des chaînes doubles brins. Cependant, si on étudie la cinétique de la réassociation, on constate que celle-ci se fait en trois phases, une fraction des fragments d’ADN se réassocie très rapidement et on arrive à un palier, puis une autre fraction d’ADN se réassocie et enfin après un certain temps on assiste à une réassociation des dernières molécules d’ADN (fig. 12). Ainsi on met en évidence trois familles de fragments d’ADN en fonction de leur vitesse de réassociation. Quelle est la signification de ces trois familles? On explique la grande vitesse de réassociation d’une partie de l’ADN par l’existence de séquences hautement répétitives, ce sont des séquences qui sont identiques à elles-mêmes et présentes en 10 5-10 6 exemplaires. Ces fragments sont localisés dans le centromère et les télomères des chromosomes; leur signification est encore discutée. Les fragments qui se réassocient après une durée moyenne sont des séquences qui sont présentes dans l’ADN en 10 2-10 4 exemplaires. Ces séquences représentent: 1o des régions régulatrices de l’ADN, régions placées en voisinage des gènes et qui en contrôlent l’expression, nous y reviendrons; 2o les régions codants pour les ARN ribosomaux et qui sont présentes en plusieurs centaines d’exemplaires; 3o certains gènes, comme ceux qui codent pour des protéines nucléaires, les histones, sont présents en grand nombre. Cette classe de séquences est appelée moyennement répétitive. La troisième et dernière classe concerne les séquences présentes en un seul ou un petit nombre d’exemplaires. Ce sont des séquences codant pour la plupart des messagers donc des protéines. Ainsi la simple étude de la vitesse de réassociation de l’ADN dénaturé, qui est en fait la probabilité de rencontre de séquences complémentaires, permet de montrer l’existence dans la molécule d’ADN de régions dont les significations sont différentes.Si l’on chauffe de l’ADN en présence d’ARN messager provenant d’un même type cellulaire et que l’on refroidit, on constate qu’il se produit une association entre l’ARN et l’ADN sur la séquence complémentaire à celle de l’ARN (U étant l’équivalent de T). La molécule formée est donc un hybride ADN-ARN. La formation d’hybrides moléculaires s’est révélée une méthode très utile pour résoudre un grand nombre de problèmes lorsque l’on est en possession d’un ARN messager ou d’une famille d’ARN messagers:– démonstration de l’existence dans une cellule d’ADN capable de donner naissance à l’ARN messager étudié;– étude par mesure cinétique de l’abondance en ARN messagers d’une préparation d’ARN;– détermination du nombre de gènes codants pour un même ARN messager.On peut par cette méthode suivre l’évolution d’un gène donné dans une cellule qui se modifie par différenciation, par cancérisation ou sous l’action de substances pharmacologiques.6. Rôle de l’acide désoxyribonucléiqueL’ADN est le support de l’information génétiqueLe fait fondamental a été établi dès 1944 par O. Avery. Celui-ci a préparé de l’ADN à partir d’une souche sauvage de pneumocoques pourvus de capsule S et a ajouté cet ADN à une souche de pneumocoques R, dépourvus de capsule. Cette addition provoque la transformation de cellule R en S, avec formation de capsule, et cette transformation devient héréditaire. Il y a donc eu transfert d’information génétique par l’ADN. Cependant cette expérience rencontra un grand scepticisme. Au cours des années cinquante, on découvrit d’autres modes de transfert de l’information par l’ADN. Dans la transduction , le transfert d’un fragment d’ADN se fait par un bactériophage qui infecte successivement deux bactéries, le bactériophage peut apporter à la seconde partie un fragment prélevé de la première. Dans la conjugaison bactérienne une cellule d’E. coli mâle peut injecter son ADN à une bactérie femelle. Il en résulte un transfert d’information d’une bactérie à l’autre. Ce transfert étant lent, on peut, en l’arrêtant à différents moments, mettre en évidence un transfert de plusieurs caractères successivement, ce qui a permis de réaliser une cartographie génétique de l’ADN de E. coli . Cependant ces expériences ne concernent que les bactéries; qu’en est-il des cellules animales? On sait que l’infection d’une cellule par un virus dont l’ADN s’incorpore dans l’ADN cellulaire va modifier les caractères de la cellule infectée. Si le virus est oncogène, la cellule prendra les caractéristiques d’une cellule cancéreuse et transmettra ces caractéristiques à sa descendance.On a réussi à transférer de l’ADN de cellules animales à d’autres cellules animales par différentes méthodes: micro-injection, incubation de la cellule réceptrice avec de l’ADN en présence de phosphate de calcium. On a ainsi provoqué la synthèse d’hémoglobine de lapin par des cellules de foie de souris, après injection d’ADN contenant le gène de l’hémoglobine de lapin à des cellules germinales de souris.Le développement des expériences de génie génétique a permis de donner des arguments indiscutables sur le rôle de l’ADN. On a pu isoler des gènes (ou synthétiser des fragments d’ADN) contenant le code d’une protéine. Le fragment d’ADN a été combiné à de l’ADN de bactérie en formant une molécule composée d’ADN bactérien et d’ADN du gène animal. Cette molécule a été réinsérée dans une bactérie. Sous certaines conditions la bactérie a synthétisé la protéine animale correspondant à l’ADN qu’elle avait reçu. On a ainsi pu faire synthétiser par E. coli de l’insuline, de l’hormone de croissance, de l’interféron humains, protéines caractérisées chimiquement et aussi par leurs propriétés biologiques.Le code génétiqueQuelle est la nature de l’information génétique contenue dans l’ADN? Le problème n’a actuellement été résolu que pour les gènes codant pour les protéines. Cela signifie que l’on a montré comment la séquence des nucléotides de l’ADN contenu dans un gène pouvait déterminer la séquence des acides aminés contenus dans la protéine codée par le gène. C’est le problème du code génétique. Le problème a été d’abord étudié par F. Crick qui a montré que trois nucléotides successifs, formant un codon ou triplet, codaient pour un acide aminé. Il existe soixante-quatre combinaisons possibles de trois nucléotides de quatre types différents, ils correspondent aux vingt acides aminés des protéines. Cela signifie que plusieurs triplets codent pour un même acide aminé. En fait trois des triplets ne codent pas pour des acides aminés mais sont placés à la fin de chaîne partie codante des gènes pour signifier la fin de la protéine. On a établi la nature des triplets qui codent pour chacun des acides aminés (M. Nirenberg, 1961-1964). Le code est universel, ce qui signifie qu’il est le même pour tous les êtres vivants, de la bactérie à l’homme. Cependant l’ADN des mitochondries, qui code pour quelques protéines et ARN mitochondriaux, utilise un code légèrement différent de celui de l’ADN nucléaire et bactérien.Organisation du génomeL’ADN est identique dans toutes les cellules d’un même organisme et cependant chacune des cellules ne synthétise pas l’ensemble des protéines synthétisées par l’ensemble de l’organisme. Par ailleurs toutes les protéines ne sont pas synthétisées en permanence. Il doit exister des mécanismes de contrôle, l’ADN doit inclure des séquences régulatrices. Celles-ci ont d’abord été décrites en 1961 par Jacob et Monod chez E. coli. Certaines régions de l’ADN contrôlent l’expression des gènes voisins en fixant l’enzyme responsable de la synthèse des ARN messagers. Beaucoup plus récemment des séquences régulatrices ont été mises en évidence dans les cellules animales. C’est ainsi que l’on a montré qu’à 30-50 nucléotides en amont de la partie 5 d’un gène il y avait une séquence appelée «boîte TATA» qui contient cette séquence tétra-nucléotidique et qui est indispensable à l’expression du gène. Un peu plus loin du gène en 5 se trouve la «boîte CAT», également indispensable. Ces séquences qui appartiennent au groupe des séquences moyennement répétitives sont donc situées en amont de la séquence codante des gènes mais sont indispensables pour que les ARN messagers correspondant à ces gènes soient synthétisés.Une autre particularité du génome, de découverte récente, est l’existence des gènes éclatés. Chaque gène qui code pour une protéine est en fait formé d’une succession de régions codantes ou exons et de régions non codantes ou introns. Le nombre d’introns et la longueur de ceux-ci varient d’un gène à l’autre mais du fait de leur existence le gène est beaucoup plus long que ne le laisserait prévoir la longueur de la protéine pour laquelle il code. Les introns existent dans la plupart des gènes animaux, et chez la levure. Ces introns vont apparaître dans l’ARN messager au moment de la synthèse de celui-ci, mais ils vont disparaître au cours de transformations qu’il subit dans le noyau. On a montré chez la levure que certains introns codaient pour des protéines qui jouent un rôle régulateur de l’expression du gène auquel ils appartiennent.Autre type de régulation, de découverte plus récente, la méthylation de l’ADN. On sait que dans l’ADN un certain nombre de cytosines étaient méthylées par une enzyme spécifique. On a montré que la méthylation de certaines cytosines provoquait l’arrêt de la synthèse d’ARN messager correspondant au gène auquel appartient la cytosine. La méthylation de l’ADN joue donc un rôle régulateur.Chromatine et expression des gènesDire qu’un gène s’exprime signifie qu’il existe une enzyme qui synthétise au contact de l’ADN des molécules d’ARN messagers, lesquelles seront traduites en protéines sur les ribosomes. Cette enzyme est l’ARN-polymérase.Il est clair que toutes les protéines d’un organisme ne sont pas synthétisées par toutes les cellules. Ainsi l’hémoglobine n’est pas synthétisée par les cellules du cerveau ou du rein. Il doit donc exister un système de sélection qui se manifestera au cours du développement fœtal et qui fera que dans un type de cellule donné certains gènes pourront s’exprimer, d’autres ne le pourront pas. L’ADN n’est pas libre, mais combiné à des protéines, le complexe ADN-protéines forme la chromatine. Certaines des protéines de la chromatine sont responsables de cette sélection des gènes au cours de la différenciation cellulaire. Par ailleurs, dans une cellule donnée, tous les gènes susceptibles de s’exprimer ne s’expriment pas nécessairement à tout moment, mais en fonction de certains signaux qui peuvent agir par exemple au niveau des séquences régulatrices de l’ADN. On rencontre, parmi les protéines de la chromatine, les enzymes nécessaires à la transcription des gènes, les enzymes qui vont modifier les histones, protéines de la chromatine, en leur fixant par exemple des radicaux phosphate ou acétyle, ce qui va influencer l’expression des gènes et la croissance cellulaire. On trouve également parmi les protéines de la chromatine des récepteurs d’hormones stéroïdes, d’hormone thyroïdienne qui vont modifier l’expression de certains gènes. La longueur de l’ADN dans la cellule est telle qu’il doit y avoir un grand état de compaction pour lui permettre de se loger dans le noyau. L’examen de la chromatine au microscope électronique révèle que l’ADN se présente sous forme d’un collier de perles, dont il serait le fil, les perles étant représentées par huit molécules d’histone. En fait l’ADN entoure le complexe d’histones, formant le nucléosome qui est l’unité morphologique de la chromatine. Le rôle des histones est en partie un rôle structural, mais également un rôle régulateur grâce à la capacité de ces protéines de fixer des radicaux. On trouve également sur les nucléosomes les protéines régulatrices que nous avons mentionnées plus haut. L’ADN seul est donc une molécule inerte et ce sont les protéines de la chromatine qui lui permettront de s’exprimer et qui contrôleront son expression.7. Rôle des acides ribonucléiquesLes ARN copient l’information génétique et la traduisent en protéines. Ce sont donc les intermédiaires de l’expression génétique.ARN messagersLes ARN messagers copient au niveau de chaque gène une chaîne d’ADN. Le produit de cette synthèse est une molécule complexe qui va subir des modifications avant de prendre sa structure définitive. La molécule d’ARN se fixe sur la partie antérieure du ribosome. Le ribosome va glisser le long du messager en donnant naissance à la chaîne protéique. Les codons se fixent successivement sur un site spécifique du ribosome. Un aminoacyl-ARN-t se combine par son anticodon au triplet du messager (fig. 13). Au fur et à mesure du glissement du ribosome, les différents aminoacyl-ARN-t viennent se fixer successivement sur les triplets d’ARN et les acides aminés se combinent les uns aux autres, permettant la formation de la protéine. La partie codante de l’ARN messager se termine toujours par un des trois codons «fin de synthèse».ARN de transfertChaque ARN de transfert se combine à un acide aminé grâce à une enzyme, l’aminoacyl-ARN-t synthétase. Il y a une spécificité stricte de chaque enzyme et de chaque ARN-t pour un acide aminé. L’anticodon des ARN-t permet à l’aminoacyl-ARN-t de se combiner au triplet caractéristique de l’acide aminé. Les ARN-t sont donc des intermédiaires obligatoires entre les acides aminés et l’ARN messager (fig. 3 et 13).ARN ribosomauxCe sont les plus abondants des ARN. Leur rôle exact est mal connu. Ils interviennent dans la structure des ribosomes et jouent un rôle dans la fixation des ARN messagers.ARN virauxCertains virus ont leur information génétique contenue non dans une molécule d’ADN, mais dans une molécule d’ARN. L’ARN de ces virus a le double rôle de support de l’information génétique et de messager de cette information sur les ribosomes cellulaires (cf. GÉNOME-Génome viral).8. Biosynthèse des acides nucléiquesDes enzymes catalysent la polymérisation des nucléotides en acides nucléiques. Les substrats de ces synthèses sont les nucléotides triphosphates, c’est-à-dire des dérivés des nucléotides comprenant trois phosphates successifs. Les deux derniers phosphates sont éliminés au moment de la polymérisation. Les nucléotides triphosphates précurseurs de l’ARN comprennent du ribose, les précurseurs de l’ADN ont du désoxyribose.Biosynthèse de l’ADNEn 1958 Meselson et Stahl, en utilisant la centrifugation en gradient de chlorure de césium et en marquant les ADN en voie de biosynthèse par un isotope lourd, 15N, montrent que la synthèse de l’ADN ou réplication est un processus semi-conservatif. Avant la division de la cellule, les deux chaînes de l’ADN se séparent et chacune sert de matrice pour la synthèse de la chaîne complémentaire, si bien que l’on aboutit à la synthèse de deux molécules identiques d’ADN, chacune de ces molécules comprenant une des chaînes initiales. Ainsi s’explique la nécessité de cette structure en double chaîne de l’ADN et le fait que des cellules provenant de la division d’une même cellule ont des ADN identiques et identiques à celui de la cellule mère. La polymérisation des nucléotides est catalysée par une enzyme, l’ADN-polymérase, étudiée par A. Kornberg. Le premier temps de la réplication est catalysé par une enzyme de déroulement qui sépare les deux chaînes. Cependant l’ADN-polymérase ne fonctionne que dans une direction, de 5 vers 3 , et nous avons vu que les deux chaînes étaient antiparallèles, donc la synthèse se fait dans des directions opposées. En fait la synthèse se fait par petits fragments d’ADN, appelés fragments d’Okazaki, qui sont synthétisés de 5 vers 3 . On a récemment montré qu’avant la synthèse de ces fragments, il se synthétise des petits fragments d’ARN qui serviront de point de départ pour la synthèse des fragments d’Okazaki. Dès que la synthèse de ces fragments s’achève, les fragments d’ARN sont détruits pour être remplacés par de l’ADN. Les fragments deviennent contigus, si bien qu’ils peuvent être joints grâce à un ADN-ligase qui assure la continuité de l’ADN (fig. 14).Il existe un autre mécanisme de synthèse de l’ADN, c’est la réparation. L’ADN peut subir des lésions, en particulier sous l’effet des radiations ultraviolettes, il peut également se produire des erreurs au cours de sa synthèse. Il existe dans les cellules un système de réparation qui agit en deux temps. Dans un premier temps la partie de la chaîne d’ADN anormale est excisée. Dans un deuxième temps elle est remplacée par un fragment d’ADN normal qui se synthétise en utilisant la chaîne normale comme matrice. Ce système de réparation est déficient dans certaines maladies héréditaires. Ces malades sont très sensibles aux brûlures solaires et ils sont très fréquemment sujets à des cancers de la peau.Biosynthèse des ARNLa synthèse de l’ARN, ou transcription , se fait au contact de l’ADN par formation de la chaîne complémentaire d’une des deux chaînes de l’ADN. La réaction peut être réalisée in vitro en mettant dans le milieu l’enzyme appelée ARN-polymérase, les quatre nucléosides triphosphates et un peu d’ADN dont une chaîne sera copiée sous forme d’ARN. In vivo , la synthèse d’ARN est précédée du déroulement d’une petite région de l’ADN qui permet de séparer sur au moins la longueur du gène les deux chaînes de l’ADN. Un problème essentiel est le mécanisme qui permet la transcription du gène entier et non pas la copie d’une région quelconque de l’ADN ne correspondant pas à un gène entier. Chez les bactéries, on a montré qu’il existait dans l’ARN-polymérase une sous-unité, appelée 靖, qui permet à l’enzyme de reconnaître une région appelée promoteur, adjacente au gène. Chez les animaux, le mécanisme de reconnaissance par l’enzyme de la partie de l’ADN correspondant au début du gène est moins bien connu. Il est possible que les boîtes TATA et CAT jouent un rôle.Cependant les ARN ne sont pas synthétisés sous leur forme définitive, mais vont subir de nombreuses modifications. Une des modifications majeures est l’élimination de la partie des ARN correspondant aux introns. En effet, l’ADN correspondant au gène est entièrement transcrit, y compris les parties qui ne seront pas traduites. Les parties de l’ARN correspondant aux introns seront éliminées par des enzymes qui sont capables de reconnaître les séquences nucléotidiques charnières et qui couperont ces séquences. Une autre enzyme combinera les uns aux autres les segments d’ARN correspondant aux exons. Ainsi aura été établie la continuité des régions codants d’ARN (cf. GÉNÉTIQUE MOLÉCULAIRE, fig. 3).Les ARN messagers comprennent à leurs parties antérieures une région non codante qui servira à la liaison de l’ARN aux ribosomes. La partie codante commence par le triplet AUG, signal de début de traduction. À la fin de l’ARN se trouve de nouveau une partie non codante qui commence nécessairement par un des triplets fin de synthèse. À l’extrême fin de la plupart des ARN messagers se synthétise, dans le noyau, une séquence poly A, grâce à une enzyme, la poly A polymérase. Cette séquence intervient vraisemblablement dans le transfert des ARN dans le cytoplasme et dans leur stabilité.Les ARN de transfert vont également subir des modifications. Après élimination de la partie correspondant aux introns va s’ajouter à la fin de la molécule une séquence C 漣C 漣A. C’est sur le A que se fixe l’acide aminé. De plus, de nombreux nucléotides vont subir des modifications: méthylations, réductions, etc. Les ARN ribosomaux sont synthétisés en une seule molécule qui va subir des coupures successives qui vont libérer les trois ARN ribosomaux.En fait les précurseurs des ARN messagers et ribosomaux ne sont jamais libres mais sont englobés dans des particules protéiques qui incluent les enzymes impliquées dans les transformations. En ce qui concerne les ARN ribosomaux, ces particules vont subir différentes transformations pour aboutir aux ribosomes définitifs.Biosynthèse des ARN dans les virus à ARNComme nous l’avons vu, certains virus ont leur information génétique contenue dans une molécule monocaténaire d’ARN. La réplication de cet ARN se fait grâce à une enzyme, l’ARN-polymérase, ARN dépendant qui permet la synthèse de la chaîne complémentaire de l’ARN viral (tabl. 3). Cette chaîne va à son tour servir de matrice pour la synthèse d’ARN complémentaire, donc identique à l’ARN viral. Ainsi le virus pourra-t-il se multiplier.Certains virus sont oncogènes, ce qui signifie que lorsqu’ils envahissent une cellule ils la transforment en cellule cancéreuse et cela de manière héréditaire. Pour les virus oncogènes à ADN, cette transformation ne peut s’effectuer que si le matériel génétique du virus s’intègre dans le matériel génétique de la cellule. En ce qui concerne ceux des virus à ARN qui sont oncogènes, une enzyme virale, la transcriptase réverse (rétrotranscriptase) va synthétiser une molécule d’ADN complémentaire de l’ARN viral, en prenant celui-ci comme matrice. Cet ADN s’insère dans l’ADN cellulaire et va s’exprimer. Il en résultera l’apparition de nouveaux caractères de la cellule, spécifiques de la transformation cancéreuse.La transcriptase réverse est donc capable de catalyser la synthèse d’ADN en utilisant l’ARN comme matrice. On l’utilise actuellement dans les expériences de génie génétique pour synthétiser l’ADN correspondant à un ARN messager spécifique d’une protéine donnée. Cet ADN est inséré dans un ADN bactérien, l’ensemble est inclus dans une bactérie et l’ADN peut dans certaines conditions synthétiser l’ARN messager et la protéine codée par cet ADN. Remarquons que l’ADN synthétisé à partir de l’ARN messager se distingue de l’ADN du gène par l’absence d’introns.
On trouve les mêmes bases que dans les ADN, à cette différence près que l’uracile remplace la thymine. Selon leur masse moléculaire et selon la fonction qu’ils assument, on distingue trois classes principales d’acides ribonucléiques:– Les acides ribonucléiques de transfert (ARN-t) sont des enchaînements de quatre-vingts nucléotides environ; ils doivent leur nom au fait qu’ils servent à transporter les acides aminés activés au cours de la synthèse des protéines. La structure primaire de plusieurs de ces molécules a été déterminée.– Les acides ribonucléiques «messagers» (ARN-m) résultent de la transcription de l’ADN par des enzymes spécifiques, les transcriptases, et constituent le code génétique utilisé par les ribosomes pour la synthèse des protéines. La masse moléculaire des ARN messagers est variable selon la longueur de la protéine qu’ils ont à synthétiser.– Divers acides ribonucléiques macromoléculaires jouent un rôle primordial: d’une part les ARN constitutifs des virus à ARN (virus de la mosaïque du tabac, de la grippe, de la poliomyélite, etc.); d’autre part, les ARN constitutifs des ribosomes, particules du cytoplasme cellulaire au niveau desquelles l’assemblage des aminoacides permet la biosynthèse des protéines.2. Structure des acides nucléiquesConformation et masse moléculaires des ARNLes ARN messagers sont des molécules filamenteuses à chaîne simple, donc monocaténaires, plus ou moins longues et de stabilité souvent précaire.Les ARN de transfert, dont on verra le rôle dans la dernière partie de cet article, sont constitués par l’enchaînement de quatre-vingts nucléotides environ, le tout ayant une masse moléculaire de l’ordre de 25 000. L’alanyl-ARN-t de levure représenté sur la figure 3 comporte soixante-dix-sept nucléotides et sa masse moléculaire est de 26 000.Dès que cette séquence et celles d’autres ARN de transfert furent connues, on se rendit compte qu’il existait une possibilité d’appariement entre les bases (stabilisant la structure de la molécule) par l’intermédiaire de liaisons non covalentes entre A 漣 U et G 漣 C (mais à l’encontre de l’ADN, le couple A 漣 U au lieu du couple A 漣 T). Plusieurs modèles ont été proposés, mais il semble que le modèle «en feuille de trèfle» rende le mieux compte des résultats expérimentaux. Des modèles de structure à trois dimensions (structure tertiaire) ont été prévus.D’autres ARN, à structure plus complexe que les ARN de transfert, c’est-à-dire par exemple l’ARN 5 S d’Escherichia coli , les ARN constitutifs des ribosomes, les ARN viraux, font également l’objet d’études assidues.Conformation et masse moléculaires de l’ADNL’ADN isolé à partir d’une source naturelle comme le thymus de veau se présente sous forme de fibres blanches et légères, comparables à première vue à de la cellulose. Sous forme de sel de sodium, l’ADN se dissout lentement dans l’eau pour former des solutions colloïdales extrêmement visqueuses.Depuis le début de ce siècle, on sait que l’ADN contient quatre bases principales: adénine (A), cytosine (C), guanine (G) et thymine (T). Le dosage de ces bases par les anciennes méthodes fit croire à leur répartition équimoléculaire, d’où la célèbre théorie des tétranucléotides qui prévalut pendant longtemps et ne fut réfutée que vers les années 1948-1950 lorsque l’on sut doser avec plus de précision les purines et les pyrimidines. Ce fut l’application extensive de la chromatographie sur papier, notamment par E. Chargaff et ses élèves, qui permit de définir un véritable atlas de la constitution en nucléotides d’un grand nombre d’acides désoxyribonucléiques naturels.La connaissance de la constitution en bases d’un ADN permet de déterminer la constitution globale de cet acide nucléique, puisque l’on sait par définition que chaque nucléotide comporte une base (à choisir parmi quatre), un pentose qui est toujours le désoxyribose et un groupement phosphoryle .Une constatation fondamentale devait se faire jour progressivement: d’une part, l’égalité des proportions en adénine et thymine et en guanine et cytosine; la constance du rapport (A + T)/(G + C) pour une même espèce animale, d’autre part. Cette constatation a pour corollaire immédiat que le rapport purines/pyrimidines est toujours égal à 1.La nature de la liaison entre nucléotides fut longtemps controversée, mais, sur la foi d’études aux rayons X et d’études enzymatiques, on s’est arrêté finalement à la liaison 3 -5 : comme on l’a déjà vu, le groupement phosphoryle établit un pont entre le groupement hydroxyle en position C-3 d’un premier nucléotide et le groupement hydroxyle en position C-5 d’un second nucléotide (fig. 2).Compte tenu des relations AT-GC, de la liaison 3 -5 -phosphodiester et des clichés obtenus aux rayons X, J. D. Watson et F. H. C. Crick proposèrent en 1952-1953 un schéma général de la conformation spatiale de l’ADN; ce schéma, mettant en évidence le rôle génétique de cet acide nucléique, est à la base de la théorie de Watson et Crick; il constitue l’un des plus grands événements scientifiques de ce siècle (fig. 4 a; cf. MA- CROMOLÉCULES).On doit se représenter la molécule d’ADN sous forme de deux chaînes hélicoïdales enroulées autour d’un même axe et maintenues sous forme d’une structure à deux brins ou structure bicaténaire (du latin catena , chaîne) grâce à l’«appariement» des bases se faisant vis-à-vis sur l’une et l’autre chaîne (fig. 4): les appariements sont toujours adénine-thymine (deux liaisons non covalentes ou liaisons hydrogène) et guanine-cytosine (trois liaisons non covalentes).Cette structure qui réunit deux chaînes réciproquement complémentaires à l’égard de leurs séquences nucléotidiques est compatible avec la réplication de la molécule d’ADN (cf. infra ).On voit sur la figure 4 b que les deux chaînes ont une polarité opposée: ce sont des chaînes antiparallèles ; de telle sorte que les extrémités 3 et 5 sont inversement placées dans chacun des deux brins. La complémentarité séquentielle et l’antiparallélisme des deux chaînes expriment le fait que leur structure est réciproquement codée .Les mesures effectuées sur la masse moléculaire des ADN ont donné lieu à de nombreuses controverses et, malgré l’énormité du travail effectué, on ne peut considérer comme valables que les mesures relatives à des échantillons dont on est sûr que l’intégrité moléculaire a été respectée. Or, une telle certitude n’est obtenue que dans les cas où l’on peut constater de visu au microscope électronique que les molécules étudiées ne sont pas rompues.Au cours des temps, diverses méthodes ont été expérimentées avec plus ou moins de succès: diffusion, diffraction de la lumière, viscosité, sédimentation. En principe, la méthode qui offre la plus grande sécurité est celle qui consiste à mesurer effectivement la longueur de molécules entières sur un cliché agrandi pris au microscope électronique (fig. 5). Connaissant d’une part l’agrandissement et sachant d’autre part que l’unité de masse moléculaire par unité de longueur pour la chaîne de l’ADN est 1,92 憐 106 par nanomètre, la masse moléculaire totale peut aisément être établie par un calcul simple, sachant que l’écart entre deux nucléotides consécutifs est de 0,34 nm. L’ultracentrifugation, effectuée sur des molécules natives, permet aussi d’obtenir d’assez bons résultats, et confirme ainsi les données précédentes.On sait actuellement que la masse moléculaire de l’ADN monocaténaire du bactériophage 淋X-174 est de 1,6 憐 106, ce qui est une très petite masse moléculaire pour un ADN contenant toute l’information nécessaire à la réplication d’un micro-organisme entier. En effet, l’ADN du virus de la vaccine comporte 240 000 paires de nucléotides, il a une masse moléculaire de 157 憐 106. Quant à l’ADN d’un micro-organisme très connu comme E. coli , il comporte 3 400 000 paires de bases représentant une masse moléculaire de 2,3 憐 109.3. Synthèse totale des acides nucléiquesLes principes fondamentaux des synthèses de nucléosides et nucléotides furent décrits par Todd et ses collaborateurs avec pour point culminant la synthèse totale de l’adénosine triphosphate, ATP en 1948. La synthèse de dinucléotides, donc l’obtention contrôlée de la liaison internucléotidique 3 , 5 -phosphodiester devait suivre grâce aux efforts indépendants de l’école de Todd et de celle de Khorana au début des années cinquante. Le perfectionnement des techniques de synthèse des oligonucléotides, couplé avec l’utilisation des méthodes enzymatiques, principalement les ADN-ligases, devait aboutir en 1976 à la synthèse totale du gène de structure régissant la biosynthèse du tyrosyl-t-ARN et en 1981 à la synthèse totale du gène interféron.Si l’application des méthodes de synthèse de l’ADN nécessite une instrumentation et une habileté exceptionnelles, en revanche les principes fondamentaux qui les régissent sont relativement simples: dans un premier temps on effectue la synthèse chimique totale d’oligonucléotides pouvant comporter de dix à quinze chaînons, et, dans un second temps, on lie entre eux ces fragments, dans l’ordre obligatoire de la séquence recherchée, grâce aux ADN-ligases; une enzyme bien connue comme l’ADN-polymérase-1 de E. coli est capable d’effectuer cette liaison.Synthèse des oligonucléotidesPlusieurs méthodes ont été successivement décrites pour l’assemblage des nucléotides par la formation réussie de la liaison 3 , 5 -phosphodiester: la méthode phosphate diester; la méthode phosphate triester; la méthode phosphite triester.Selon la méthode phosphite triester qui semble la plus prometteuse à l’heure actuelle, on effectue la liaison 3 , 5 -phosphodiester en faisant réagir deux nucléosides convenablement protégés avec un dichloroalkyl-phosphite (fig. 6 a). Le dinucléoside monophosphite obtenu est oxydé par l’iode en solution aqueuse pour donner le dinucléoside monophosphate correspondant. Cette méthode représente un progrès appréciable par rapport aux méthodes antérieures utilisant des phosphates substitués, en particulier quant à la rapidité.Un autre progrès récent dans la synthèse des oligonucléotides réside dans la transposition de la méthode sur support solide. Le principe de cette méthode est le suivant (fig. 6 b): la première unité A de la chaîne est attachée à un matériel macroscopique tel que du polystyrène ou des billes de silice. La seconde unité B et tous les agents de condensation sont dissous dans un solvant approprié et mélangés avec le support solide. La réaction de condensation se produit entre B et A sur le support solide et la chaîne se trouve étendue à S 漣 A 漣 B. À la fin de la période de réaction, le surplus des réactifs et les solvants sont éliminés par lavage. On renouvelle l’opération autant de fois que le nécessite l’élongation de la chaîne. Avec une telle méthode, il est impératif que les rendements soient aussi près que possible de 100 p. 100 à chacune des étapes, sinon le rendement final après dix ou quinze étapes chute de façon verticale: à titre d’exemple, si le rendement de condensation à chaque étape est de 90 p. 100, au bout de dix étapes le rendement final n’est que de 35 p. 100.Cette méthode est automatisée, de telle sorte que la synthèse d’oligonucléotides comportant jusqu’à quatorze unités nucléotidiques soit maintenant un procédé de routine. La cadence est de l’ordre de 30 minutes par chaînon; le système ne nécessite pas d’expérience préalable de la part de l’utilisateur; il fonctionne jour et nuit sans avoir besoin de surveillance, jusqu’à la terminaison du programme qui a été introduit au début de l’expérience.De plus, le même principe s’applique aussi à la synthèse de l’ARN!...Assemblage des oligonucléotides et obtention de gènes synthétiquesL’assemblage des oligonucléotides obtenus par synthèse chimique totale est réalisé par liaison enzymatique au moyen d’ADN-ligases. Dans un exemple tel que celui représenté par la figure 6, on voit que deux icosanucléotides partiellement appariés forment un assemblage bicaténaire suffisamment rigide pour que l’on puisse y accrocher, grâce à une ADN-ligase, un oligonucléotide de la même longueur que les deux premiers et qui viendra lui-même s’apparier sur une partie de sa longueur. L’allongement progressif de la chaîne se fait donc par accrochage successif d’oligonucléotides synthétiques de séquence connue.L’une des performances les plus remarquables dans ce domaine est la synthèse totale du gène de l’interféron leucocytaire humain IFN- 見-1 par Edge et ses collaborateurs (1981). L’interféron IFN- 見-1 est une protéine comportant 166 résidus d’aminoacides. Ce gène exprimé par E. coli programme la synthèse d’un interféron en tous points identique au produit naturel (De Maeyer et al., 1982). Il s’agit d’un fragment d’ADN bicaténaire comportant 514 paires de nucléotides. La synthèse a comporté la fabrication préliminaire de 66 oligonucléotides dont la dimension s’échelonne entre 14 et 21 résidus, plus un dodécanucléotide, le tout par des procédés en phase solide. La liaison de ces oligonucléotides fut obtenue au moyen de l’ADN-ligase du phage T 4.Mais bien entendu, pour pouvoir reconstituer des molécules biologiquement actives, il est indispensable de connaître préalablement la séquence selon laquelle l’assemblage des nucléotides doit être réalisé: c’est cette analyse que nous exposerons maintenant.Détermination des séquences et de la structure primaire de l’ADNLa détermination de la séquence complète des nucléotides constitutifs d’ADN macromoléculaires biologiquement actifs représente une des performances les plus remarquables de ces dernières années. Un premier succès important fut remporté par Sanger et ses collaborateurs (1975) avec la détermination de la séquence complète des nucléotides constitutifs de l’ADN monocaténaire circulaire du phage 淋X-174, soit 5 375 nucléotides. Par la suite ces mêmes auteurs devaient perfectionner leur méthode par l’emploi d’inhibiteurs dits «terminateurs de chaînes» ce qui leur a permis de déterminer la séquence complète de l’ADN bicaténaire d’un organisme supérieur: il s’agit en fait du génome mitochondrial humain, soit 16 569 paires de bases (Anderson et al., 1981). Ces travaux considérables devaient valoir à Sanger l’attribution du prix Nobel de chimie, pour la seconde fois, en 1980.Méthode de SangerLe principe de cette analyse est le suivant: dans un premier temps on coupe l’ADN à étudier en certains points grâce aux enzymes adéquates, les nucléases, qui se caractérisent par leur remarquable spécificité d’action (tabl. 2), notamment les endonucléases de restriction [cf. GÉNIE GÉNÉTIQUE].On obtient de cette façon un certain nombre de fragments dits oligonucléotidiques; on sépare, et on purifie, ces fragments par électrophorèse sur gel de polyacrylamide. On détermine la séquence de chacun des fragments et on reconstitue la séquence originelle en juxtaposant tous les segments dûment identifiés.La découverte de nucléotides substitutifs dits terminateurs de chaîne a permis de séquencer les fragments d’ADN avec une grande sûreté. À partir d’un fragment monocaténaire dit chaîne 漣, qui jouera le rôle de matrice , on induit la synthèse d’une chaîne complémentaire +, sous l’action de l’ADN-polymérase-1, mais on peut bloquer l’allongement (élongation) de cette chaîne + grâce aux terminateurs.Le principe du fonctionnement des terminateurs de chaîne est illustré (fig. 7) par l’exemple du 2 , 3 -didéoxythymidine-5 -triphosphate (ddTTP) sur l’ADN-polymérase-1 lorsque ce composé est incorporé dans la chaîne nucléotidique croissante au lieu et place de l’acide thymidylique. L’absence sur ce ddTT du groupement hydroxyle en position C-3 stoppe l’élongation de la chaîne polynucléotidique à partir de l’endroit où normalement T aurait dû être incorporé. Si l’on incube une amorce (un court fragment d’ADN nécessaire pour amorcer la réaction de synthèse) avec l’ADN-polymérase-1 et une matrice (ADN monocaténaire inconnu) avec un mélange de ddTTP, de TTP (thymidine triphosphate normal) et des trois autres nucléosides triphosphates normaux dont l’un est marqué avec 32 P, l’ADN-polymérase catalyse la formation d’une série de chaînes complémentaires de la matrice; on obtient finalement un mélange de polynucléotides ayant tous la même séquence initiale et terminés par des groupements ddT à l’extrémité C-3 . Lorsque l’on fractionne ce mélange par électrophorèse sur gel de polyacrylamide, le diagramme de résolution montre toutes les bandes indiquant la distribution de ddT (fig. 8).En utilisant des terminateurs du même type pour les autres nucléotides dans des mélanges d’incubation séparés et en mettant les incubats en parallèle sur le gel d’électrophorèse, on obtient un diagramme de résolution dont on peut lire la séquence «comme dans un livre».On peut utiliser deux types de triphosphates terminateurs, les dérivés didésoxy et les arabinosides. Les ara-nucléoside-triphosphates fonctionnent comme des terminateurs de chaînes inhibiteurs de l’ADN-polymérase-1 de E. coli d’une façon comparable à ddTP bien que les chaînes se terminant par ara-C puissent être étendues par certaines ADN-polymérases de mammifères (cette propriété explique la valeur thérapeutique des arabinosides qui bloquent, par exemple, la réplication des ADN viraux en respectant la réplication des ADN des cellules hôtes: cf. THÉRAPEUTIQUE - Chimiothérapie). Afin d’avoir une résolution suffisante des bandes permettant une lecture de séquences étendues, il est obligatoire que la proportion de triphosphate terminateur par rapport au triphosphate normal soit telle que l’on ait une incorporation partielle du terminateur: il est évident que si l’on avait 100 p. 100 de terminateur, toutes les chaînes se termineraient avec l’incorporation de la première molécule d’analogue.Pour les didésoxy-nucléoside-triphosphates, la proportion est de 1 pour 100. Pour les ara-nucléoside-triphosphates, la proportion est de 1 pour 5 000.4. Propriétés physico-chimiquesUtilisation des radiations dans l’analyse des acides nucléiquesLes acides nucléiques présentent en solution diluée un très intense spectre d’absorption en ultraviolet ; ce spectre est la résultante des spectres d’absorption individuels des purines et pyrimidines entrant dans leur constitution. Du point de vue quantitatif, il est intéressant car il permet de doser les acides nucléiques en solution pure (fig. 9).Les méthodes utilisant la diffraction des rayons X fournissent des informations capitales en ce qui concerne la structure des molécules possédant un degré d’organisation élevé, ce qui est le cas des macromolécules d’intérêt biologique telles que les protéines cristallisées, les acides nucléiques macromoléculaires, les virus. Toutefois, la visualisation des structures à trois dimensions n’est pas directe et ne peut être obtenue que par une interprétation laborieuse des clichés photographiques de diffraction. On trouvera dans l’article BIOLOGIE MOLÉCULAIRE l’histoire de la découverte de la structure tridimensionnelle de l’ADN par W. T. Atsbury puis Wilkins, Watson et Crick.En revanche, la microscopie électronique permet une observation directe des macromolécules convenablement préparées; bien qu’elle nécessite un appareil expérimental onéreux et des spécialistes hautement qualifiés, cette technique est cependant d’une interprétation beaucoup plus accessible.Dans le cas des acides nucléiques, on doit d’abord isoler les molécules dans un état aussi voisin que possible de l’état natif pour éviter les fragmentations, puis les fixer par un ombrage métallique; on obtient alors des clichés très intéressants, qui ont permis non seulement d’avoir une idée de la conformation des acides nucléiques macromoléculaires, mais encore de mesurer la dimension exacte des molécules (fig. 10).CentrifugationL’ADN peut être fractionné par centrifugation en gradient de chlorure de césium, CsCl. Le césium a un poids atomique très élevé: 133. Si l’on soumet une solution de chlorure de césium 8 M à centrifugation rapide pendant trois jours, on constate qu’il s’établit un gradient linéaire de concentration correspondant à un gradient de densité entre 1,55 g/cm3 au sommet du tube et 1,80 g/cm3 au fond du tube. On peut dissoudre un mélange d’ADN dans la solution de chlorure de césium et centrifuger. Après soixante-douze heures, les différents ADN formeront des bandes à différents niveaux. On dit que la densité apparente d’une molécule d’ADN est la densité de chlorure de césium au niveau duquel l’ADN sédimentera en fin de centrifugation à l’équilibre. La densité apparente d’un ADN est proportionnelle au pourcentage de G-C qui le compose. On peut ainsi séparer différents ADN. On a également utilisé cette méthode pour séparer un ADN préalablement marqué par des isotopes lourds (15N, 2H) du même ADN non marqué. Les ARN extraits d’une cellule peuvent être fractionnés par centrifugation dans un gradient de saccharose linéaire entre 5 et 20 p. 100. Ce gradient est préformé dans le tube à centrifuger et on dépose à la surface le mélange d’ARN en solution. Après dix-huit heures de centrifugation à 50 000 g , on retrouve la plupart des ARN séparés (on les détecte en expulsant lentement le liquide du tube et en mesurant en continu la densité optique à 260 nm). Cette méthode peu résolutive permet de séparer les grands ARN ribosomaux et les ARN de transfert 4 S (fig. 10).ÉlectrophorèseL’électrophorèse en gel de polyacrylamide permet de séparer les macromolécules en fonction de leur charge et de leur poids moléculaire. Les acides nucléiques sont des polyanions comprenant la même charge par nucléotide, la séparation se fera seulement en fonction de leur longueur (et dans une certaine mesure de leur conformation).Nous avons vu l’application de cette méthode à la détermination de la séquence de l’ADN, sachant que la vitesse de migration électrophorétique de chaque fragment est inversement proportionnelle au logarithme de sa longueur.La même méthode appliquée à un mélange d’ARN permet de séparer les ARN 28 S, 18 S, 5 S, 4 S ainsi que dans certains cas les ARN messagers, lorsque ceux-ci sont suffisamment abondants et de taille homogène (fig. 10).5. Dénaturation de l’ADN. Hybrides moléculairesLorsque l’on chauffe une solution d’ADN, on constate que son absorption d’ultraviolet à 260 nanomètres augmente de 40 p. 100 à partir d’une certaine température, c’est l’effet hyperchromique . La température correspondant à la moitié de l’effet hyperchromique est appelée température de fusion ou Tm. L’effet hyperchromique correspond à une rupture des liaisons hydrogène entre les deux chaînes d’ADN. La séparation entre les deux chaînes avec perte de la structure secondaire de l’ADN est appelée dénaturation thermique de l’ADN (fig. 11).On peut par un traitement mécanique casser l’ADN en des fragments de longueurs plus ou moins homogènes. On chauffe le mélange au-dessus de la température de fusion, puis on refroidit progressivement. On constate alors que les chaînes complémentaires se réassocient en reformant des chaînes doubles brins. Cependant, si on étudie la cinétique de la réassociation, on constate que celle-ci se fait en trois phases, une fraction des fragments d’ADN se réassocie très rapidement et on arrive à un palier, puis une autre fraction d’ADN se réassocie et enfin après un certain temps on assiste à une réassociation des dernières molécules d’ADN (fig. 12). Ainsi on met en évidence trois familles de fragments d’ADN en fonction de leur vitesse de réassociation. Quelle est la signification de ces trois familles? On explique la grande vitesse de réassociation d’une partie de l’ADN par l’existence de séquences hautement répétitives, ce sont des séquences qui sont identiques à elles-mêmes et présentes en 10 5-10 6 exemplaires. Ces fragments sont localisés dans le centromère et les télomères des chromosomes; leur signification est encore discutée. Les fragments qui se réassocient après une durée moyenne sont des séquences qui sont présentes dans l’ADN en 10 2-10 4 exemplaires. Ces séquences représentent: 1o des régions régulatrices de l’ADN, régions placées en voisinage des gènes et qui en contrôlent l’expression, nous y reviendrons; 2o les régions codants pour les ARN ribosomaux et qui sont présentes en plusieurs centaines d’exemplaires; 3o certains gènes, comme ceux qui codent pour des protéines nucléaires, les histones, sont présents en grand nombre. Cette classe de séquences est appelée moyennement répétitive. La troisième et dernière classe concerne les séquences présentes en un seul ou un petit nombre d’exemplaires. Ce sont des séquences codant pour la plupart des messagers donc des protéines. Ainsi la simple étude de la vitesse de réassociation de l’ADN dénaturé, qui est en fait la probabilité de rencontre de séquences complémentaires, permet de montrer l’existence dans la molécule d’ADN de régions dont les significations sont différentes.Si l’on chauffe de l’ADN en présence d’ARN messager provenant d’un même type cellulaire et que l’on refroidit, on constate qu’il se produit une association entre l’ARN et l’ADN sur la séquence complémentaire à celle de l’ARN (U étant l’équivalent de T). La molécule formée est donc un hybride ADN-ARN. La formation d’hybrides moléculaires s’est révélée une méthode très utile pour résoudre un grand nombre de problèmes lorsque l’on est en possession d’un ARN messager ou d’une famille d’ARN messagers:– démonstration de l’existence dans une cellule d’ADN capable de donner naissance à l’ARN messager étudié;– étude par mesure cinétique de l’abondance en ARN messagers d’une préparation d’ARN;– détermination du nombre de gènes codants pour un même ARN messager.On peut par cette méthode suivre l’évolution d’un gène donné dans une cellule qui se modifie par différenciation, par cancérisation ou sous l’action de substances pharmacologiques.6. Rôle de l’acide désoxyribonucléiqueL’ADN est le support de l’information génétiqueLe fait fondamental a été établi dès 1944 par O. Avery. Celui-ci a préparé de l’ADN à partir d’une souche sauvage de pneumocoques pourvus de capsule S et a ajouté cet ADN à une souche de pneumocoques R, dépourvus de capsule. Cette addition provoque la transformation de cellule R en S, avec formation de capsule, et cette transformation devient héréditaire. Il y a donc eu transfert d’information génétique par l’ADN. Cependant cette expérience rencontra un grand scepticisme. Au cours des années cinquante, on découvrit d’autres modes de transfert de l’information par l’ADN. Dans la transduction , le transfert d’un fragment d’ADN se fait par un bactériophage qui infecte successivement deux bactéries, le bactériophage peut apporter à la seconde partie un fragment prélevé de la première. Dans la conjugaison bactérienne une cellule d’E. coli mâle peut injecter son ADN à une bactérie femelle. Il en résulte un transfert d’information d’une bactérie à l’autre. Ce transfert étant lent, on peut, en l’arrêtant à différents moments, mettre en évidence un transfert de plusieurs caractères successivement, ce qui a permis de réaliser une cartographie génétique de l’ADN de E. coli . Cependant ces expériences ne concernent que les bactéries; qu’en est-il des cellules animales? On sait que l’infection d’une cellule par un virus dont l’ADN s’incorpore dans l’ADN cellulaire va modifier les caractères de la cellule infectée. Si le virus est oncogène, la cellule prendra les caractéristiques d’une cellule cancéreuse et transmettra ces caractéristiques à sa descendance.On a réussi à transférer de l’ADN de cellules animales à d’autres cellules animales par différentes méthodes: micro-injection, incubation de la cellule réceptrice avec de l’ADN en présence de phosphate de calcium. On a ainsi provoqué la synthèse d’hémoglobine de lapin par des cellules de foie de souris, après injection d’ADN contenant le gène de l’hémoglobine de lapin à des cellules germinales de souris.Le développement des expériences de génie génétique a permis de donner des arguments indiscutables sur le rôle de l’ADN. On a pu isoler des gènes (ou synthétiser des fragments d’ADN) contenant le code d’une protéine. Le fragment d’ADN a été combiné à de l’ADN de bactérie en formant une molécule composée d’ADN bactérien et d’ADN du gène animal. Cette molécule a été réinsérée dans une bactérie. Sous certaines conditions la bactérie a synthétisé la protéine animale correspondant à l’ADN qu’elle avait reçu. On a ainsi pu faire synthétiser par E. coli de l’insuline, de l’hormone de croissance, de l’interféron humains, protéines caractérisées chimiquement et aussi par leurs propriétés biologiques.Le code génétiqueQuelle est la nature de l’information génétique contenue dans l’ADN? Le problème n’a actuellement été résolu que pour les gènes codant pour les protéines. Cela signifie que l’on a montré comment la séquence des nucléotides de l’ADN contenu dans un gène pouvait déterminer la séquence des acides aminés contenus dans la protéine codée par le gène. C’est le problème du code génétique. Le problème a été d’abord étudié par F. Crick qui a montré que trois nucléotides successifs, formant un codon ou triplet, codaient pour un acide aminé. Il existe soixante-quatre combinaisons possibles de trois nucléotides de quatre types différents, ils correspondent aux vingt acides aminés des protéines. Cela signifie que plusieurs triplets codent pour un même acide aminé. En fait trois des triplets ne codent pas pour des acides aminés mais sont placés à la fin de chaîne partie codante des gènes pour signifier la fin de la protéine. On a établi la nature des triplets qui codent pour chacun des acides aminés (M. Nirenberg, 1961-1964). Le code est universel, ce qui signifie qu’il est le même pour tous les êtres vivants, de la bactérie à l’homme. Cependant l’ADN des mitochondries, qui code pour quelques protéines et ARN mitochondriaux, utilise un code légèrement différent de celui de l’ADN nucléaire et bactérien.Organisation du génomeL’ADN est identique dans toutes les cellules d’un même organisme et cependant chacune des cellules ne synthétise pas l’ensemble des protéines synthétisées par l’ensemble de l’organisme. Par ailleurs toutes les protéines ne sont pas synthétisées en permanence. Il doit exister des mécanismes de contrôle, l’ADN doit inclure des séquences régulatrices. Celles-ci ont d’abord été décrites en 1961 par Jacob et Monod chez E. coli. Certaines régions de l’ADN contrôlent l’expression des gènes voisins en fixant l’enzyme responsable de la synthèse des ARN messagers. Beaucoup plus récemment des séquences régulatrices ont été mises en évidence dans les cellules animales. C’est ainsi que l’on a montré qu’à 30-50 nucléotides en amont de la partie 5 d’un gène il y avait une séquence appelée «boîte TATA» qui contient cette séquence tétra-nucléotidique et qui est indispensable à l’expression du gène. Un peu plus loin du gène en 5 se trouve la «boîte CAT», également indispensable. Ces séquences qui appartiennent au groupe des séquences moyennement répétitives sont donc situées en amont de la séquence codante des gènes mais sont indispensables pour que les ARN messagers correspondant à ces gènes soient synthétisés.Une autre particularité du génome, de découverte récente, est l’existence des gènes éclatés. Chaque gène qui code pour une protéine est en fait formé d’une succession de régions codantes ou exons et de régions non codantes ou introns. Le nombre d’introns et la longueur de ceux-ci varient d’un gène à l’autre mais du fait de leur existence le gène est beaucoup plus long que ne le laisserait prévoir la longueur de la protéine pour laquelle il code. Les introns existent dans la plupart des gènes animaux, et chez la levure. Ces introns vont apparaître dans l’ARN messager au moment de la synthèse de celui-ci, mais ils vont disparaître au cours de transformations qu’il subit dans le noyau. On a montré chez la levure que certains introns codaient pour des protéines qui jouent un rôle régulateur de l’expression du gène auquel ils appartiennent.Autre type de régulation, de découverte plus récente, la méthylation de l’ADN. On sait que dans l’ADN un certain nombre de cytosines étaient méthylées par une enzyme spécifique. On a montré que la méthylation de certaines cytosines provoquait l’arrêt de la synthèse d’ARN messager correspondant au gène auquel appartient la cytosine. La méthylation de l’ADN joue donc un rôle régulateur.Chromatine et expression des gènesDire qu’un gène s’exprime signifie qu’il existe une enzyme qui synthétise au contact de l’ADN des molécules d’ARN messagers, lesquelles seront traduites en protéines sur les ribosomes. Cette enzyme est l’ARN-polymérase.Il est clair que toutes les protéines d’un organisme ne sont pas synthétisées par toutes les cellules. Ainsi l’hémoglobine n’est pas synthétisée par les cellules du cerveau ou du rein. Il doit donc exister un système de sélection qui se manifestera au cours du développement fœtal et qui fera que dans un type de cellule donné certains gènes pourront s’exprimer, d’autres ne le pourront pas. L’ADN n’est pas libre, mais combiné à des protéines, le complexe ADN-protéines forme la chromatine. Certaines des protéines de la chromatine sont responsables de cette sélection des gènes au cours de la différenciation cellulaire. Par ailleurs, dans une cellule donnée, tous les gènes susceptibles de s’exprimer ne s’expriment pas nécessairement à tout moment, mais en fonction de certains signaux qui peuvent agir par exemple au niveau des séquences régulatrices de l’ADN. On rencontre, parmi les protéines de la chromatine, les enzymes nécessaires à la transcription des gènes, les enzymes qui vont modifier les histones, protéines de la chromatine, en leur fixant par exemple des radicaux phosphate ou acétyle, ce qui va influencer l’expression des gènes et la croissance cellulaire. On trouve également parmi les protéines de la chromatine des récepteurs d’hormones stéroïdes, d’hormone thyroïdienne qui vont modifier l’expression de certains gènes. La longueur de l’ADN dans la cellule est telle qu’il doit y avoir un grand état de compaction pour lui permettre de se loger dans le noyau. L’examen de la chromatine au microscope électronique révèle que l’ADN se présente sous forme d’un collier de perles, dont il serait le fil, les perles étant représentées par huit molécules d’histone. En fait l’ADN entoure le complexe d’histones, formant le nucléosome qui est l’unité morphologique de la chromatine. Le rôle des histones est en partie un rôle structural, mais également un rôle régulateur grâce à la capacité de ces protéines de fixer des radicaux. On trouve également sur les nucléosomes les protéines régulatrices que nous avons mentionnées plus haut. L’ADN seul est donc une molécule inerte et ce sont les protéines de la chromatine qui lui permettront de s’exprimer et qui contrôleront son expression.7. Rôle des acides ribonucléiquesLes ARN copient l’information génétique et la traduisent en protéines. Ce sont donc les intermédiaires de l’expression génétique.ARN messagersLes ARN messagers copient au niveau de chaque gène une chaîne d’ADN. Le produit de cette synthèse est une molécule complexe qui va subir des modifications avant de prendre sa structure définitive. La molécule d’ARN se fixe sur la partie antérieure du ribosome. Le ribosome va glisser le long du messager en donnant naissance à la chaîne protéique. Les codons se fixent successivement sur un site spécifique du ribosome. Un aminoacyl-ARN-t se combine par son anticodon au triplet du messager (fig. 13). Au fur et à mesure du glissement du ribosome, les différents aminoacyl-ARN-t viennent se fixer successivement sur les triplets d’ARN et les acides aminés se combinent les uns aux autres, permettant la formation de la protéine. La partie codante de l’ARN messager se termine toujours par un des trois codons «fin de synthèse».ARN de transfertChaque ARN de transfert se combine à un acide aminé grâce à une enzyme, l’aminoacyl-ARN-t synthétase. Il y a une spécificité stricte de chaque enzyme et de chaque ARN-t pour un acide aminé. L’anticodon des ARN-t permet à l’aminoacyl-ARN-t de se combiner au triplet caractéristique de l’acide aminé. Les ARN-t sont donc des intermédiaires obligatoires entre les acides aminés et l’ARN messager (fig. 3 et 13).ARN ribosomauxCe sont les plus abondants des ARN. Leur rôle exact est mal connu. Ils interviennent dans la structure des ribosomes et jouent un rôle dans la fixation des ARN messagers.ARN virauxCertains virus ont leur information génétique contenue non dans une molécule d’ADN, mais dans une molécule d’ARN. L’ARN de ces virus a le double rôle de support de l’information génétique et de messager de cette information sur les ribosomes cellulaires (cf. GÉNOME-Génome viral).8. Biosynthèse des acides nucléiquesDes enzymes catalysent la polymérisation des nucléotides en acides nucléiques. Les substrats de ces synthèses sont les nucléotides triphosphates, c’est-à-dire des dérivés des nucléotides comprenant trois phosphates successifs. Les deux derniers phosphates sont éliminés au moment de la polymérisation. Les nucléotides triphosphates précurseurs de l’ARN comprennent du ribose, les précurseurs de l’ADN ont du désoxyribose.Biosynthèse de l’ADNEn 1958 Meselson et Stahl, en utilisant la centrifugation en gradient de chlorure de césium et en marquant les ADN en voie de biosynthèse par un isotope lourd, 15N, montrent que la synthèse de l’ADN ou réplication est un processus semi-conservatif. Avant la division de la cellule, les deux chaînes de l’ADN se séparent et chacune sert de matrice pour la synthèse de la chaîne complémentaire, si bien que l’on aboutit à la synthèse de deux molécules identiques d’ADN, chacune de ces molécules comprenant une des chaînes initiales. Ainsi s’explique la nécessité de cette structure en double chaîne de l’ADN et le fait que des cellules provenant de la division d’une même cellule ont des ADN identiques et identiques à celui de la cellule mère. La polymérisation des nucléotides est catalysée par une enzyme, l’ADN-polymérase, étudiée par A. Kornberg. Le premier temps de la réplication est catalysé par une enzyme de déroulement qui sépare les deux chaînes. Cependant l’ADN-polymérase ne fonctionne que dans une direction, de 5 vers 3 , et nous avons vu que les deux chaînes étaient antiparallèles, donc la synthèse se fait dans des directions opposées. En fait la synthèse se fait par petits fragments d’ADN, appelés fragments d’Okazaki, qui sont synthétisés de 5 vers 3 . On a récemment montré qu’avant la synthèse de ces fragments, il se synthétise des petits fragments d’ARN qui serviront de point de départ pour la synthèse des fragments d’Okazaki. Dès que la synthèse de ces fragments s’achève, les fragments d’ARN sont détruits pour être remplacés par de l’ADN. Les fragments deviennent contigus, si bien qu’ils peuvent être joints grâce à un ADN-ligase qui assure la continuité de l’ADN (fig. 14).Il existe un autre mécanisme de synthèse de l’ADN, c’est la réparation. L’ADN peut subir des lésions, en particulier sous l’effet des radiations ultraviolettes, il peut également se produire des erreurs au cours de sa synthèse. Il existe dans les cellules un système de réparation qui agit en deux temps. Dans un premier temps la partie de la chaîne d’ADN anormale est excisée. Dans un deuxième temps elle est remplacée par un fragment d’ADN normal qui se synthétise en utilisant la chaîne normale comme matrice. Ce système de réparation est déficient dans certaines maladies héréditaires. Ces malades sont très sensibles aux brûlures solaires et ils sont très fréquemment sujets à des cancers de la peau.Biosynthèse des ARNLa synthèse de l’ARN, ou transcription , se fait au contact de l’ADN par formation de la chaîne complémentaire d’une des deux chaînes de l’ADN. La réaction peut être réalisée in vitro en mettant dans le milieu l’enzyme appelée ARN-polymérase, les quatre nucléosides triphosphates et un peu d’ADN dont une chaîne sera copiée sous forme d’ARN. In vivo , la synthèse d’ARN est précédée du déroulement d’une petite région de l’ADN qui permet de séparer sur au moins la longueur du gène les deux chaînes de l’ADN. Un problème essentiel est le mécanisme qui permet la transcription du gène entier et non pas la copie d’une région quelconque de l’ADN ne correspondant pas à un gène entier. Chez les bactéries, on a montré qu’il existait dans l’ARN-polymérase une sous-unité, appelée 靖, qui permet à l’enzyme de reconnaître une région appelée promoteur, adjacente au gène. Chez les animaux, le mécanisme de reconnaissance par l’enzyme de la partie de l’ADN correspondant au début du gène est moins bien connu. Il est possible que les boîtes TATA et CAT jouent un rôle.Cependant les ARN ne sont pas synthétisés sous leur forme définitive, mais vont subir de nombreuses modifications. Une des modifications majeures est l’élimination de la partie des ARN correspondant aux introns. En effet, l’ADN correspondant au gène est entièrement transcrit, y compris les parties qui ne seront pas traduites. Les parties de l’ARN correspondant aux introns seront éliminées par des enzymes qui sont capables de reconnaître les séquences nucléotidiques charnières et qui couperont ces séquences. Une autre enzyme combinera les uns aux autres les segments d’ARN correspondant aux exons. Ainsi aura été établie la continuité des régions codants d’ARN (cf. GÉNÉTIQUE MOLÉCULAIRE, fig. 3).Les ARN messagers comprennent à leurs parties antérieures une région non codante qui servira à la liaison de l’ARN aux ribosomes. La partie codante commence par le triplet AUG, signal de début de traduction. À la fin de l’ARN se trouve de nouveau une partie non codante qui commence nécessairement par un des triplets fin de synthèse. À l’extrême fin de la plupart des ARN messagers se synthétise, dans le noyau, une séquence poly A, grâce à une enzyme, la poly A polymérase. Cette séquence intervient vraisemblablement dans le transfert des ARN dans le cytoplasme et dans leur stabilité.Les ARN de transfert vont également subir des modifications. Après élimination de la partie correspondant aux introns va s’ajouter à la fin de la molécule une séquence C 漣C 漣A. C’est sur le A que se fixe l’acide aminé. De plus, de nombreux nucléotides vont subir des modifications: méthylations, réductions, etc. Les ARN ribosomaux sont synthétisés en une seule molécule qui va subir des coupures successives qui vont libérer les trois ARN ribosomaux.En fait les précurseurs des ARN messagers et ribosomaux ne sont jamais libres mais sont englobés dans des particules protéiques qui incluent les enzymes impliquées dans les transformations. En ce qui concerne les ARN ribosomaux, ces particules vont subir différentes transformations pour aboutir aux ribosomes définitifs.Biosynthèse des ARN dans les virus à ARNComme nous l’avons vu, certains virus ont leur information génétique contenue dans une molécule monocaténaire d’ARN. La réplication de cet ARN se fait grâce à une enzyme, l’ARN-polymérase, ARN dépendant qui permet la synthèse de la chaîne complémentaire de l’ARN viral (tabl. 3). Cette chaîne va à son tour servir de matrice pour la synthèse d’ARN complémentaire, donc identique à l’ARN viral. Ainsi le virus pourra-t-il se multiplier.Certains virus sont oncogènes, ce qui signifie que lorsqu’ils envahissent une cellule ils la transforment en cellule cancéreuse et cela de manière héréditaire. Pour les virus oncogènes à ADN, cette transformation ne peut s’effectuer que si le matériel génétique du virus s’intègre dans le matériel génétique de la cellule. En ce qui concerne ceux des virus à ARN qui sont oncogènes, une enzyme virale, la transcriptase réverse (rétrotranscriptase) va synthétiser une molécule d’ADN complémentaire de l’ARN viral, en prenant celui-ci comme matrice. Cet ADN s’insère dans l’ADN cellulaire et va s’exprimer. Il en résultera l’apparition de nouveaux caractères de la cellule, spécifiques de la transformation cancéreuse.La transcriptase réverse est donc capable de catalyser la synthèse d’ADN en utilisant l’ARN comme matrice. On l’utilise actuellement dans les expériences de génie génétique pour synthétiser l’ADN correspondant à un ARN messager spécifique d’une protéine donnée. Cet ADN est inséré dans un ADN bactérien, l’ensemble est inclus dans une bactérie et l’ADN peut dans certaines conditions synthétiser l’ARN messager et la protéine codée par cet ADN. Remarquons que l’ADN synthétisé à partir de l’ARN messager se distingue de l’ADN du gène par l’absence d’introns.
Encyclopédie Universelle. 2012.
